kierownik pracowni:
dr inż. Michał Marczyk
W ramach pracowni prowadzone są projekty w obszarach tematycznych:
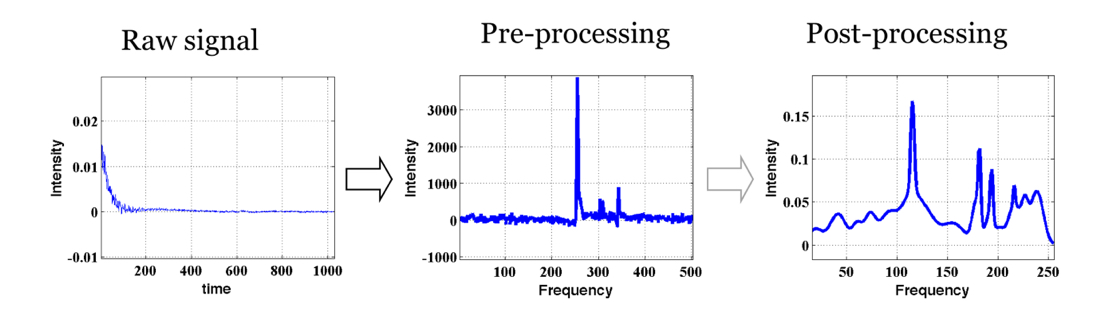
Opracowanie i implementacja algorytmów analizy widm spektroskopii magnetycznego rezonansu jądrowego.
W trakcie przeprowadzania prac w podanej tematyce zaobserwowano że bardzo duży wpływ na jakość finalnego wyniku, tj. wyznaczania stężeń metabolitów zależy bardzo silnie od przeprowadzonego wstępnego przetworzenia danych. Po wnikliwiej analizie zaobserwowano że możliwa jest propozycja technik automatycznego strojenia parametrów dla pięciu powszechnie używanych metod korekty fazy (problemu który występuje w analizie widm NMR): Automics. metoda minimalizacji entropii. metoda Ernsta, metoda eDispa oraz Dispa. Zaproponowane modyfikacje i techniki strojenia zaowocowały poprawą jakości widma i zmniejszeniem błędu estymacji składu chemicznego badanej tkanki o co najmniej 5% co jest wynikiem bardzo dobrym. W celu analizy ilościowej widma zaproponowano algorytm dekompozycji widma częstotliwościowego do modelu mieszanin Gaussowskich. W modelu tym każdy pik widma reprezentowany jest przez składową modelu. Podejście to pozwala na wyznaczenie informacji o piku w danym widmie nawet w przypadku gdy jest on złączony z innymi pikami. Zaproponowana metodyka została zweryfikowana eksperymentalnie na zbiorze widm pozyskanych z fantomu dla którego znany był skład chemiczny. Proponowana metoda analizy sygnału skutkuje uzyskaniem bardzo precyzyjnego wyniku. Przeprowadzono analizę porównawczą pomiędzy proponowaną metodyką a metodami takimi jak: LC MODEL i Tarquin. W wyniku tego porównania zaobserwowano że proponowana metodyka skutkuje wynikiem na porównywalnym poziomie precyzji lecz jest bardziej uniwersalna gdyż nie wymaga posiadania dodatkowych baz wiedzy i może służyć dla celów analizy dowolnego widma. W opinii autorów jest to jedna z większych zalet opracowanej metody. Wynikiem powyżej opisanych prac jest program GNMR (opracowany w środowisku MATLAB) służący do automatycznej analizy danych spektroskopii magnetycznego rezonansu jądrowego.
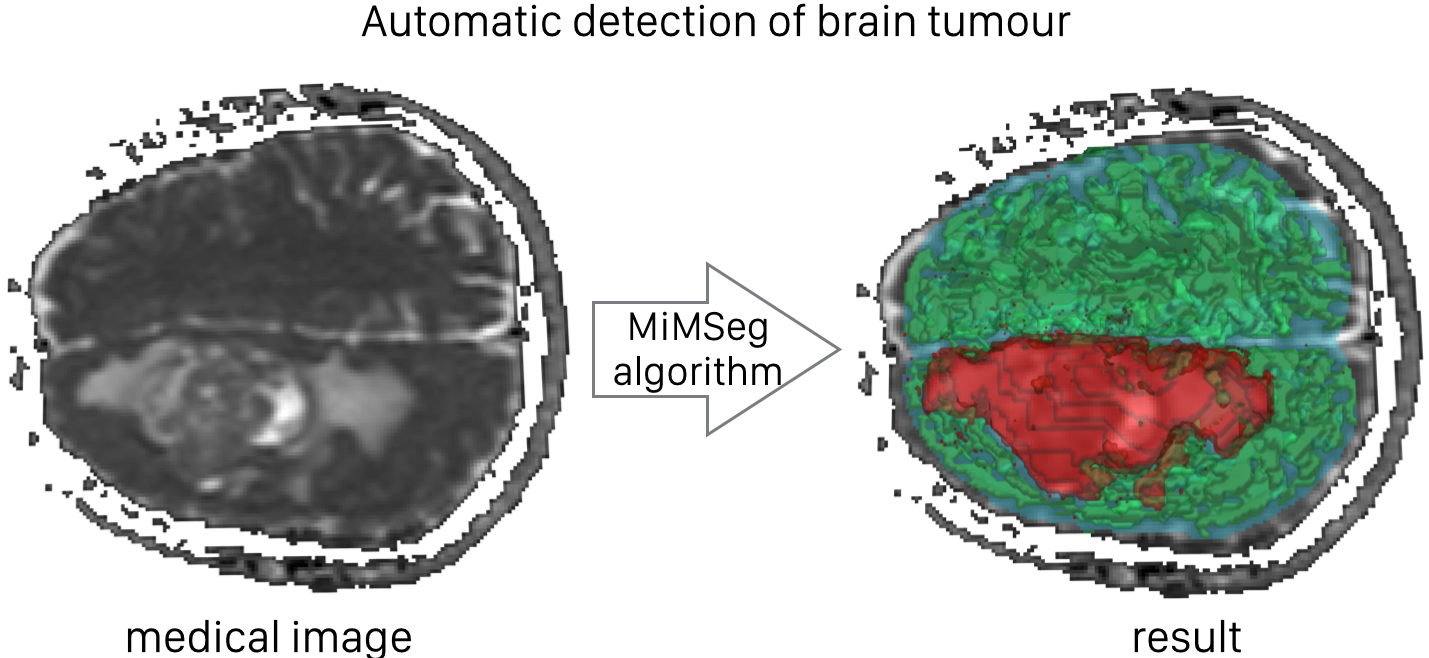
Opracowanie algorytmu automatycznej segmentacji guzów mózgu z wykorzystaniem obrazów zależnych od dyfuzji (DWI).
Celem tych była propozycja algorytmu pozwalającego na w pełni automatyczna detekcję guza mózgu glioblastoma multiforme z wykorzystaniem obrazowania zależnego od dyfuzji. W technice tej uzyskiwana jest informacja o dyfuzji cząsteczek wody w badanej tkance (w przestrzeni zewnątrzkomórkowej) co pozwala na detekcję różnego typu tkanek które charakteryzowane są przez różną ilość różnego typu komórek. Jedną z wielkości która może zostać wyznaczona jest tak zwany pozorny współczynnik dyfuzji (ADC). W wyniku prac zaproponowano algorytm pozwalający na estymację charakterystycznego progu odcięcia wartości pozornego współczynnika dyfuzji dla różnego typu tkanek. Opracowane rozwiązanie oparte jest o dekompozycję rozkładu sygnału ADC do matematycznego modelu mieszanin Gaussowskich a następnie zgrupowaniu komponent z wykorzystaniem algorytmu k-średnich. Walidację proponowanego algorytmu przeprowadzono z wykorzystaniem zestawu danych zebranych przed operacją dla 17 pacjentów z rozpoznaniem guza gliablastoma multiforme. W wyniku analizy opartej o schemat walidacji krzyżowej „leave one out” uzyskano wynik automatycznej segmentacji zgodny w 90% (wyrażone indeksem Dice’a) z segmentacją manualną, co porównując do najnowszych metod prezentowanych na arenie międzynarodowej (konkurs BRATS organizowany przez stowarzyszenie MICAII) jest wynikiem bardzo dobrym. Opracowane narzędzie znajduje się obecnie w fazie publikacji i przygotowania programu z interfejsem przyjaznym użytkownikowi.
Kolejnym krokiem realizowanym w ramach tego tematu było opracowanie metody automatycznej segmentacji guza glioblastoma na podstawie tzw. częściowej segmentacji. Rozwiązanie to ma na celu przyśpieszenie pracy lekarza poprzez detekcję guza tylko na podstawie niewielkiego jego obszaru który może zostać zdefiniowany bardzo szybko. Zaproponowano wykorzystanie 16 sekwencji obrazowania magnetycznego rezonansu jądrowego. Jako aparat matematyczny wybrano maszynę wektorów podpierających. Uzyskano wynik równy 86%.
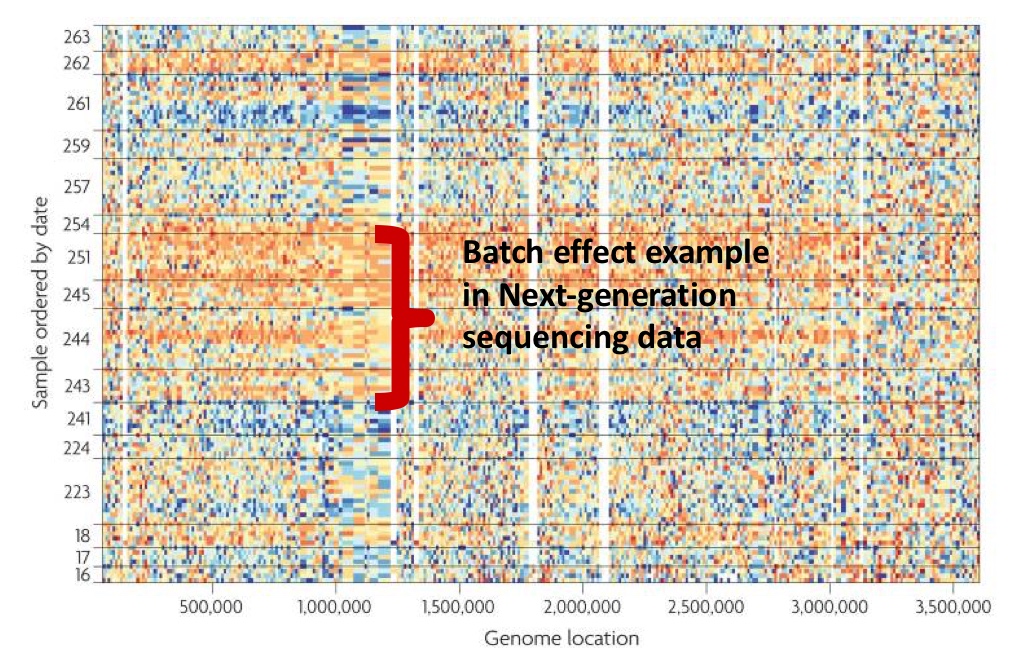
Opracowanie i implementacja algorytmów korekcji tzw. efektu paczki w danych pozyskanych z wykorzystaniem mikromacierzy DNA.
Efekt paczki może być zdefiniowany jako wpływ źródeł technicznej zmienności występującej w danych będących wynikiem badań biologii molekularnej o wysokiej przepustowości. Efekt ten powodują nieoczekiwane różnice w danych, które są wynikiem wielu czynników związanych ze zwykle z etapem przygotowania eksperymentu, Co więcej niektóre z tych czynników są niemożliwe do przewidzenia. W wyniku przeprowadzonych prac opracowano metodę eliminacji tego efektu poprzez podział danych na poszczególne podgrupy (paczki) z wykorzystaniem techniki programowania dynamicznego. Uzyskane wstępne wyniki analiz wskazują, że odpowiedni dobór procedur identyfikacji efektu paczki odgrywa kluczową rolę w analizie wyników eksperymentów biologicznych.
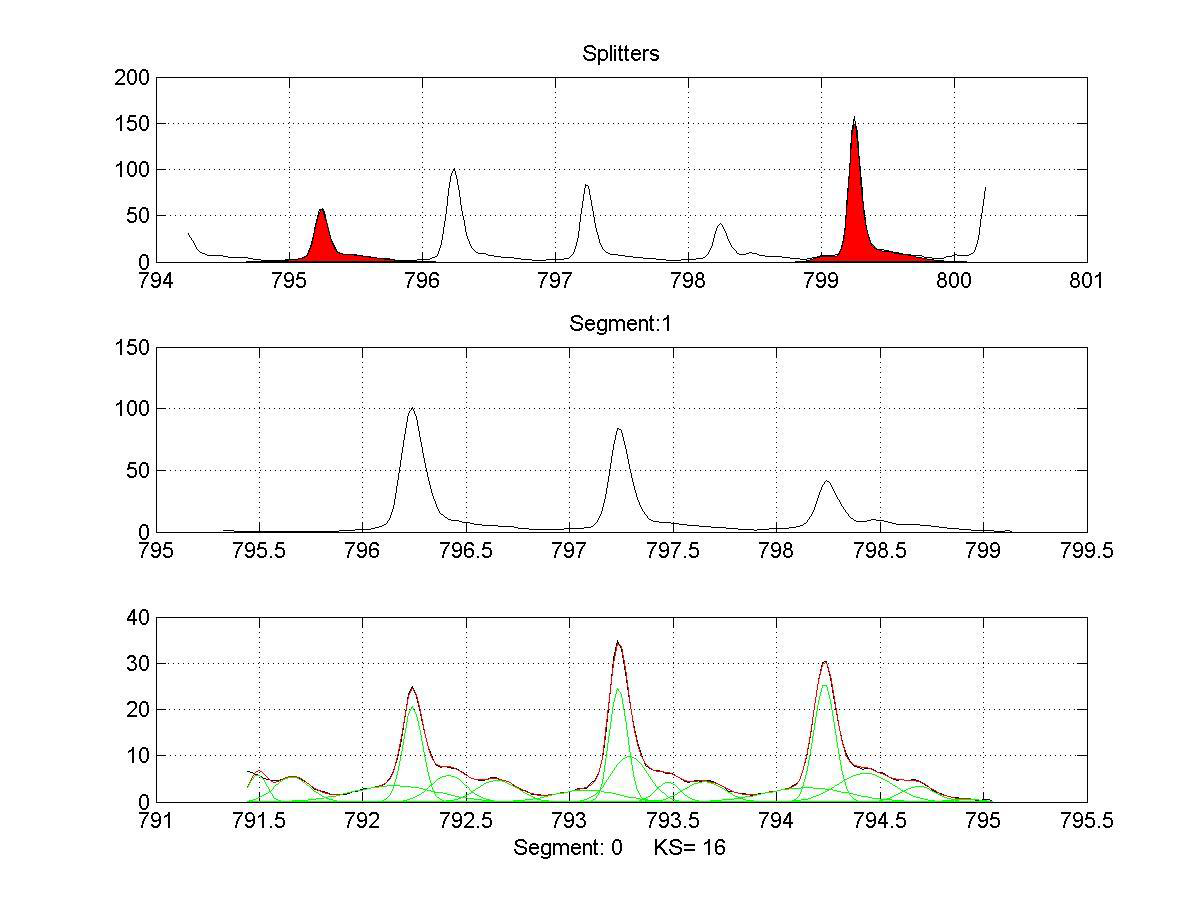
Opracowanie algorytmów przetwarzania i analizy widm spektrometrii MALDI TOF, w celach poszukiwania biomarkerów choroby nowotworowej.
W ramach projektu stworzono kompletne narzędzie analizy białkowych widm masowych oparte o wydajny algorytm poszukiwania warunków początkowych dla jednowymiarowego modelu mieszanin gaussowskich. Stworzona metodologia została zastosowana do analizy danych niskiej rozdzielczości tj. widm MALDI-ToF oraz danych wysokiej rozdzielczości, takich jak widma obrazowania molekularnego IMS. Ponadto w celu przyspieszenia obliczeń wprowadzono procedury programowania równoległego z wykorzystaniem wielu rdzeni procesora oraz kart graficznych wykorzystujących technologię CUDA. Dwuwymiarowy model mieszanin normalnych został również użyty do analizy obrazów żeli dwukierunkowej elektroforezy w celu efektywnego wykrywania plam białkowych.
Analiza danych genomowych pod kątem odpowiedzi na pytanie czy dawki promieniowania jonizującego wpływają na zmianę ilości rekombinacji genów typu V.
W trakcie prac badano czy dawka promieniowania i czas który upływa po napromienieniu wpływają na częstotliwość rekombinacji typu V w genomie. Jako materiał badawczy wykorzystano informację z eksperymentu przeprowadzonego na różnego typu liniach komórkowych myszy.
Analiza i implementacja metod integracji danych w celu zwiększenia mocy eksperymentów biologicznych.
Zespół przy współpracy z Public Health England (UK) i King Faisal Hospital (Saudi Arabia), zajmuje się problematyką wskazania genetycznego podłoża radiowrażliwości (radiation response) – indywidualnej reakcji na promieniowanie jonizujące. W tym calu wykonano studia typu GWAS na populacji o zróżnicowanym stopniu pokrewieństwa, gdzie bliźnięta służą jako grypa walidująca wyniki uzyskane na poziomie osób niespokrewnionych. Badania te mają interesujące znaczenie dla rozwoju zindywidualizowanej radioterapii, gdzie to kombinacja polimorfizmów może być jednym z głównych czynników dostosowania dawki leczniczej. W trakcie realizacji badań wykorzystano metody integracji prawdopodobieństw otrzymanych w wyniku testowania zróżnicowania interakcji SNP-gen. Ponieważ studium prowadzone jest na małej populacji metody te pozwoliły na zwiększenie mocy prowadzonych obliczeń przy jednoczesnym zachowaniu informacji biologicznej, a nawet jej zwiększeniu.
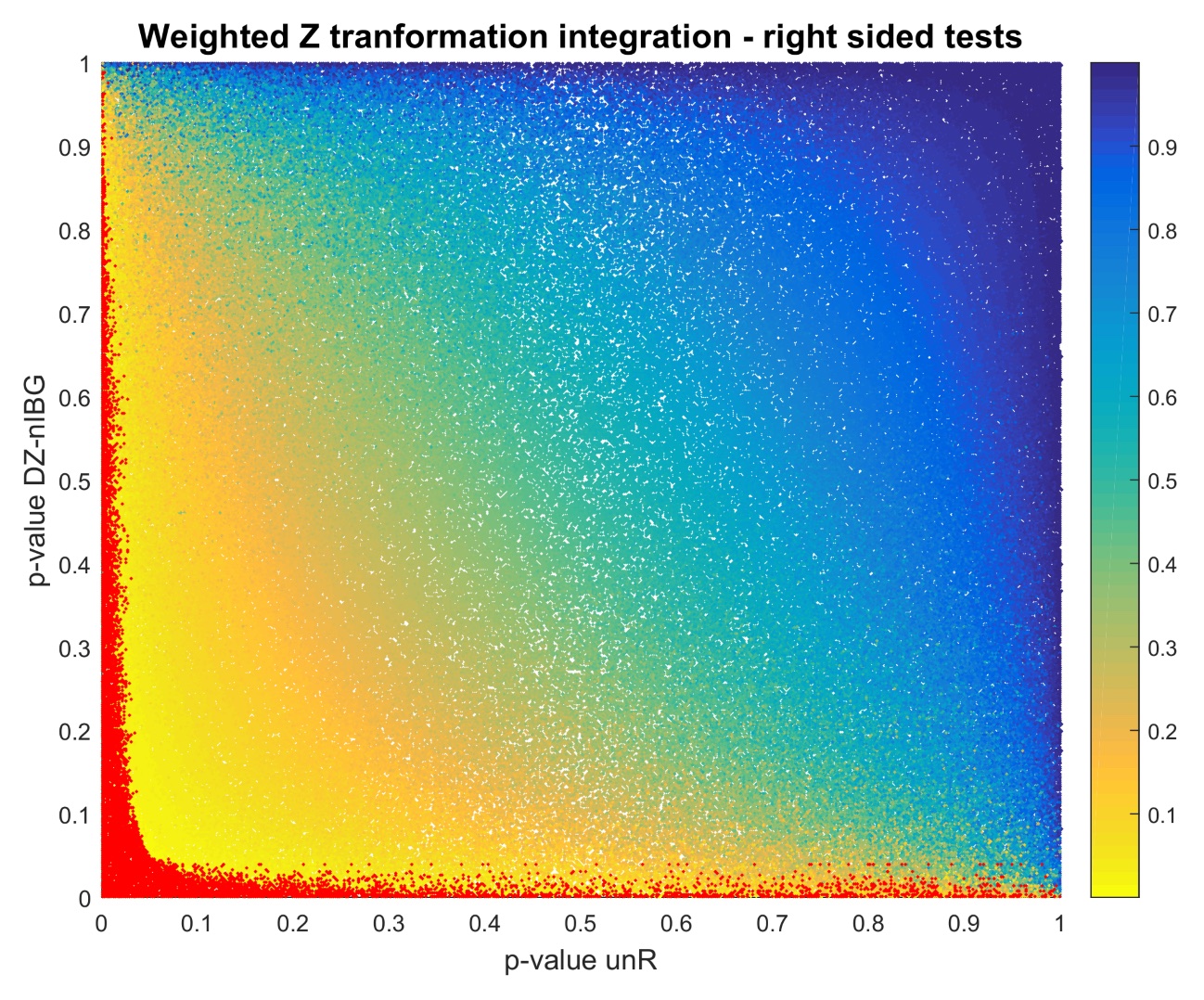
Poszukiwanie regionów o zmienionej metylacji pomiędzy typami białaczki u ludzi.
Prace w tym obszarze badań koncentrują się na analizie ilościowej genów ulegających metylacji . Zaimplementowano metody wstępnego przetwarzania danych polegające na znormalizowaniu poziomu metylacji, od 0 (brak metylacji) do 1 (pełna metylacja). Kolejnym krokiem przygotowania danych do analizy było wdrożenie miary tzw. M-value pokazującej poziom metylacji, log2 (zmetylowane / niezmetylowane). Miarę tę cechuje przede wszystkim brak ograniczeń zakresu co jest niewątpliwą wadą innych stosowanych miar.
Poszukiwanie biomarkerów choroby nowotworowej z wykorzystaniem wyników obrazowania molekularnego.
Dla danych ze spektroskopii molekularnej MALDI-MSI przeprowadzono analizę nienadzorowaną: wykorzystujemy dostosowany algorytm k-średnich by poszukiwać obszarów heterogenicznych pod względem sygnatury molekularnej. Najpierw widmo średnie dekomponowane jest na mieszaninę rozkładów gaussowskich, następnie dokonuje się filtracji szumów poprzez filtrację cech nisko amplitudowych. Później rekurencyjnie, aż do osiągnięcia żądanego stopnia zagnieżdżenia, dokonujemy sekcji cech charakterystycznych dla danego podobszaru i szukany jest podział, który jest wskazywany przed indeks Dunna jako optymalny. Przetwarzane były do tej pory dane z raka głowy i szyi, aktualnie obliczenia skupiają się na raku tarczycy.
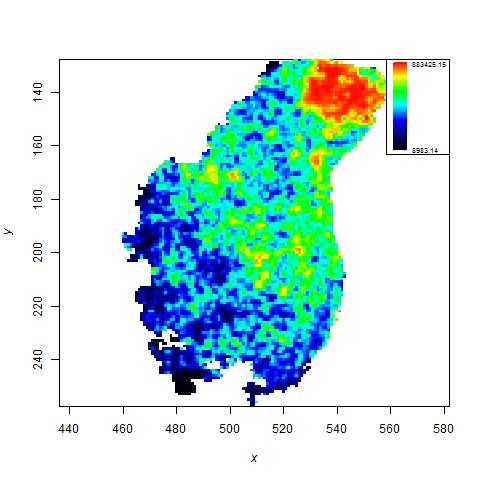
Analiza zmian o charakterze CNV w genomie (copy number variation) dla celów oceny radiowrażliwości.
Radiowrażliwość jest określana jako względna wrażliwość komórek, tkanek, narządów lub organizmów na szkodliwe działanie promieniowania jonizującego. Inny poziom promienioczułości jest głównym powodem różnic wpływu radioterapii i jej skutków ubocznych wśród pacjentów. Jest to szczególnie ważna kwestia w przypadku gdy możliwe jest spersonalizowanie radioterapii. Niektóre badania wskazują, że indywidualna odporność na czynniki środowiskowe może być spowodowana istnieniem zmian CNV w DNA. Celem badań było opracowanie metodyki analizy zmian w strukturze genomu w zależności od dawki promieniowania jonizującego. Badano ponadto wpływ różnego stopnia radiowrażliwości różnych linii komórkowych na obserwowane różne efekty wpływu promieniowania.
Badania prowadzone były we współpracy z wieloma ośrodkami zagranicznymi i krajowymi takimi jak: Centrun Onkologii w Gliwicach, Public Health England, King Fasail Hospital w Arabii Saudyjskiej, CEA Grenoble oraz DKFZ Heidelberg.
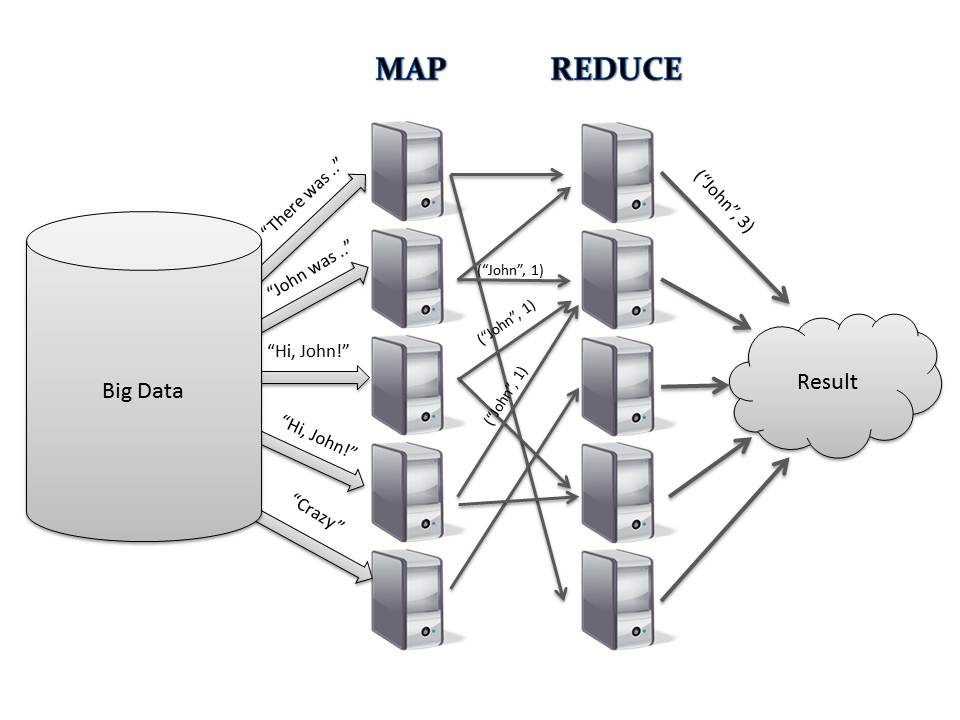
Modyfikacja metody selekcji cech „Monte Carlo” w oparciu o zastosowanie technik obliczeń rozproszonych i programowania równoległego.
Zaproponowano rozwiązanie problemu dużej złożoności obliczeniowej i długiego czasu obliczeń poprzez zastosowanie techniki obliczeń rozproszonych HADOOP i elementów programowania równoległego na każdym etapie przetwarzania danych. Zaproponowana architektura programu pozwala na skalowanie problemu do wielu maszyn w klastrach komputerowych co potencjalnie umożliwia nie tylko przyśpieszenie analizy lecz także na pracę równoległą wielu użytkowników (np. 1-5). W wyniku prac przeprowadzonych w ramach tematu 10 opracowano: Podstawową wersję strony internetowej pozwalającej na kontrolowanie opracowanej aplikacji bez potrzeby dodatkowego oprogramowania klienta (z możliwością przesłania zestawu danych na serwer. Otwarto również pole do pracy z dużymi danymi bez potrzeby ich składowania w pamięci maszyn obliczeniowych.
Analiza epidemiologiczna występowania cukrzycy typu I wśród dzieci w Polsce.
W pracy analizowano zapadalność na cukrzycę typu I wśród dzieci w województwie Śląskim w porównaniu z innymi regionami kraju oraz niektórymi krajami europejskimi. Zaproponowano szereg rozwiązań metodologicznych w wyniku czego opracowano szereg skryptów pozwalających na analizę danych epidemiologicznych. W wyniku prac dowiedziono iż najszybszy wzrost zapadalności wystąpił u dzieci w średnim wieku (5-9 lat). Nie zaobserwowano istotnej różnicy dla poszczególnych płci. Całkowita częstość występowania wzrosła o 380% sugerujące epidemię cukrzycy typu 1 u dzieci i młodzieży na Śląsku. Te zmiany czasowe potwierdzają, że Polska ma obecnie jeden z najwyższych wskaźników zachorowalności na cukrzycę typu 1 u dzieci w Europie.





